
蘑菇的DNA序列可以用于物种鉴定、系统发育和进化分子钟等分析。
物种鉴定是利用蘑菇基因组中称为“DNA条形码”的特定序列,通过测序DNA条形码并和数据库中已知物种的序列进行比对,来鉴定物种。DNA条形码是指在同一个物种内相似,在不同的物种间存在区别,从而能用于鉴定物种的特定DNA片段。除此之外,DNA条形码一般还具有以下特征:序列较短,容易扩增;在不同种类的真菌中都存在。鉴定蘑菇常用的DNA条形码序列包括ITS和LSU序列。
系统发育是基于通过不同的算法,分析不同蘑菇的特定基因序列,推测其分化过程并构建系统发育树。真菌中常用的系统发育算法包括最大简约法(Maximum Parsimony,MP)、最大似然法(Maximum Likehood,ML)和贝叶斯法(Bayes)。
系统发育树可以分为无根树和有根树。无根树仅表示物种之间分化关系的远近,并不知道谁更早分化;有根树则是在无根树“置根”之后得到的,可以显示物种分化的先后顺序。置根通常采用外类群法——在树中引入一个外类群,其亲缘关系足够远,必定是树中最早分化出来的,基于该外类群推测其他所有剩余类群的分化关系,也可以采用中点法——取分化最远的两个枝条的中点为根。
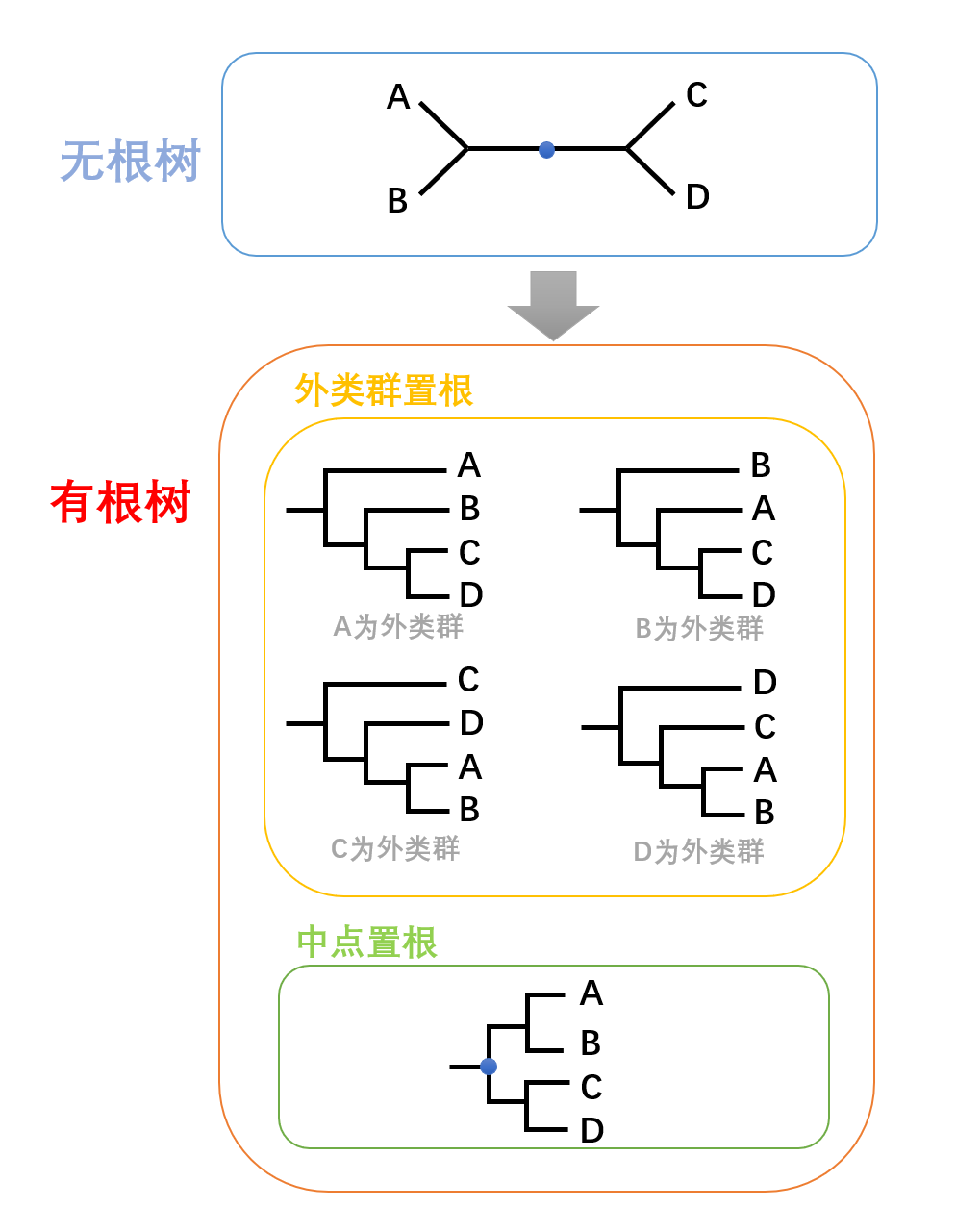
根据枝长的含义,有根树还可以进一步分为cladogram(枝条长度无意义,整个树仅显示系统发育的结构)、chronogram(枝条长度表征分化时间)和phylogram(枝条长度表征序列的变化程度)。
为了检验系统发育树的可靠程度,可以采用一些算法(如bootstrap法)来计算各个分支的自展支持率,该数值一般会标在树枝上,单位为%但百分号常省略不写,一般大于50或70被认为是可接受的。
构建系统发育树可以采用多种序列,最常用的还是ITS和LSU,也可以用SSU、β-tubulin、rbp1、rbp2或tef1DNA序列,有时甚至可以用蛋白质序列建树。一般来说,研究近缘物种,要用变化快(不保守)的序列,因为保守序列在近缘物种内往往都很相似;研究远缘物种,要用变化慢(保守)的序列,因为不保守序列常常会变化到无法比较的程度。
分子钟是利用DNA序列推断物种分化时间的一种方法。其理论基础是分子进化的中性理论,该理论认为,同一个基因的演化速度是相对恒定的,因此我们可以通过已知的化石记录确定某一个基因的演化速度,再通过物种之间基因的差异程度倒推物种的分化程度。
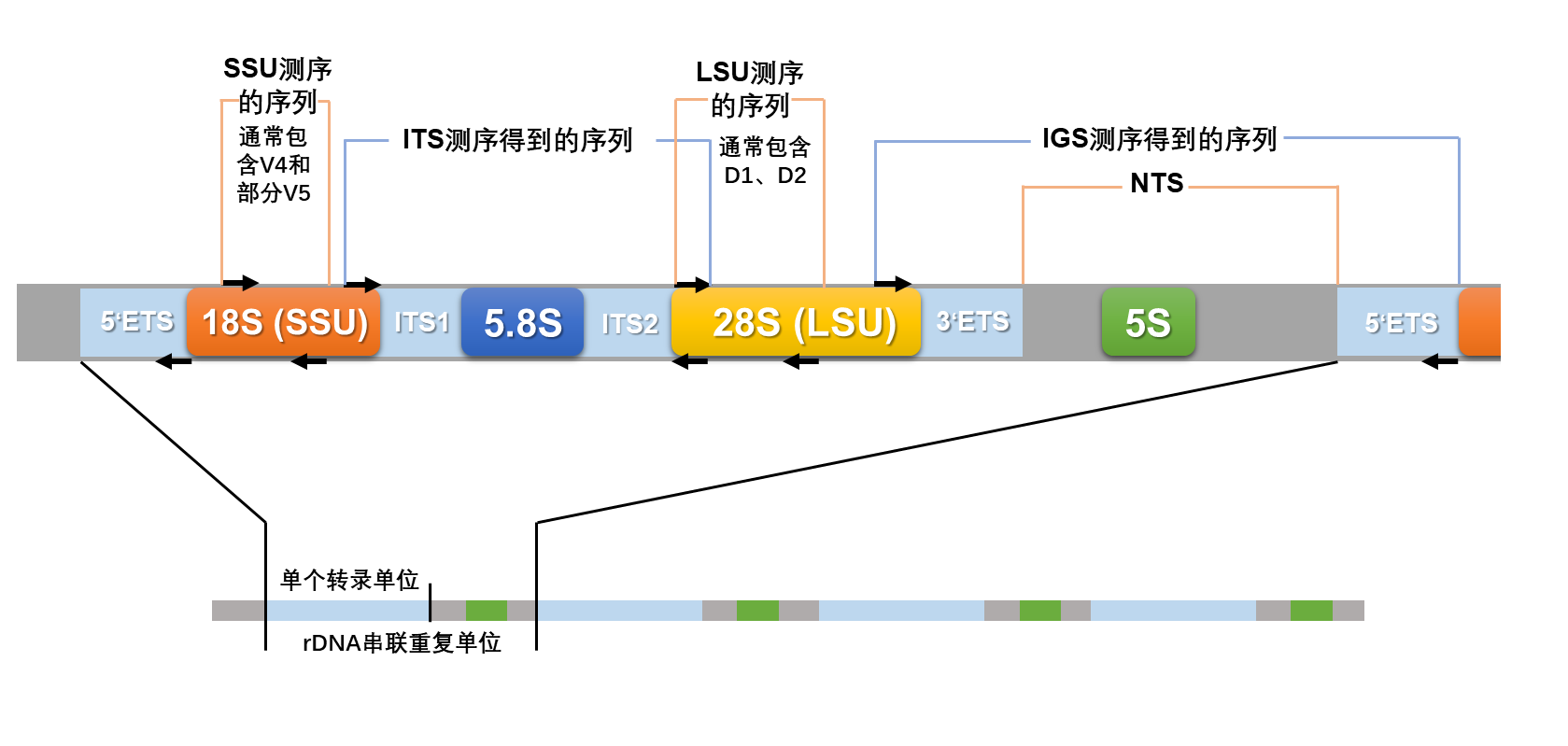
典型的高等真菌细胞核核糖体DNA(rDNA)结构示意图。
真菌的rDNA由18S(橙)、5.8S(深蓝)、28S(黄,实际根据物种的不同,可能为25-28S)和5S(绿)四种组成。它们在基因组内按上述顺序排列并重复多次出现。其中,前三者在转录时作为同一个转录单位(图中淡蓝色区域)被转录,而5S rDNA则被独立转录。
18S rDNA转录产物构成核糖体的小亚基,因此被称为核糖体小亚基基因序列(SSU),剩下三者共同构成核糖体大亚基,但一般仅有28S rDNA被称为核糖体大亚基基因序列(LSU)。在18S、5.8S和28S基因之间的片段称为ITS序列。注意,实际研究中测序的SSU和LSU序列通常不完整,而获得的ITS序列往往还包括一部分的SSU、完整5.8S和一部分LSU序列。
除了上述序列,转录单位(淡蓝色)的头和尾都有ETS序列。在一个转录单位和下一个转录单位之间的部分称作NTS序列,NTS包含5S rDNA基因。NTS、ETS统称为IGS序列。

 下载完成后,可以用记事本打开.gb或.fasta文件。GenBank格式的内容和网页上展示的内容是一样的。Fasta格式的文件如下:
下载完成后,可以用记事本打开.gb或.fasta文件。GenBank格式的内容和网页上展示的内容是一样的。Fasta格式的文件如下:>NR_151745.1 Agaricus campestris MA Fungi:80998 ITS region; from TYPE material
GGAAGGATCATTATTGAATTATGTTTCTAGATGGGTTGTAGCTGGCTCTTTGGAGCATGTGCACACCTGT
TTGGATTTCATTTTCATCCACCTGTGCACCTATTGTAGTCTTTGGTTTGGGTATTGAGGAAGTGGTCAGC
CTATCAGCATTTGCTGGATGTGAGGAGTTTGCAGTGTGAAAGCATTGCTGTCCTTTACTTGGCCATGGAG
TCTTTTGCCTACCAGAGTCTATGTCATTCATTATACCCTGTCGAATGTTATCGAATGTCTTTACATGGGC
TTTCATGCCTATGAAAATTATAATACAACTTTCAGCAACGGATCTCTTGGCTCTCGCATCGATGAAGAAC
GCAGCGAAATGCGATAAGTAATGTGAATTGCAGAATTCAGTGAATCATCGAATCTTTGAACGCATCTTGC
GCTCCTTGGTACTCCGAGGAGCATGCCTGTTTGAGTGTCATTAAATTCTCAACTCTCTTATACTTTGTTG
TATAGGAGGGCTTGGATTGTGGAGGTTTGCTGGCAACTTGTTTGTGGTCAGCTCCTCTGAAATGCATTAG
CGGAACCGTTTGCGATCTGCCACAAGTGTGATAAACTATCTACACTGGCGAGGGGATTGCTCTCTGTGTT
TGTTCAGCTTCTAATCGTCTCAGTTTGAGACAACTTTTGAATACTTGACCTCANATCAGGTAGGACTACC
CGCTGAACTTAA